python の可視化ツールは matplotlib が有名ですがそのプラグイン的役割を果たすモジュールとして seaborn があります。seaborn でプロットすると勝手に回帰直線をプロットしてくれたりするのですが今回は seaborn.clustermap を取り上げます。
seaborn.clustermap とは
seaborn.clustermap は入力された二次元データフレームを行と列それぞれでクラスタリングし、近いクラスタ同士を近い場所に配置するよう並び替えを行った後 heatmap + デンドログラム(樹形図)として可視化することができる関数です。
API: https://seaborn.pydata.org/generated/seaborn.clustermap.html
適当にあやめデータセットでやると以下のようになります。それっぽい図が出てきて楽しい気分になります。
import seaborn as sns df_iris = sns.load_dataset('iris') species = df_iris.pop("species") g = sns.clustermap(df_iris) g.savefig('clustermap.png', dpi=150)

一方でこのクラスタリングがどういうロジックに基づいて動いているのか、カスタマイズするときはどうやったらええのか、ベストプラクティス的なことはないのか、など気になる点があるのでそれについてまとめて行きます。
ロジックについて
clustermap ではクラスタリングをすべて scipy.cluster.hierarchy.linkage にまかしているという記述があります。実際 clustermap 関数を呼び出すと
ClusterClass インスタンスが新しく生成され、インスタンスの plot メソッドが呼ばれる- plot 内部で
self.plot_dendgramが呼ばれる self.dendgramではdendgrammethod が呼ばれ内部で_DendrogramPlotterインスタンスが新しく生成され、インスタンスの plot が呼び出される- Dendgram.plot で scipy.cluster.hierarchy.linkage によって linkage が生成される
という流れで処理が走ります。というわけでまずは scipy の linkage を理解する必要が有ることがわかりました。
Scipy.cluster.hierarchy.linkage
linkage は階層型クラスタリングを行う関数です。
Scipy.cluster.hierarchy.linkage API scipy.cluster.hierarchy.linkage — SciPy v1.1.0 Reference Guide
具体的には特定の指標のベクトル $y$ を入力としてその hierarchy linkage を返します。 階層型クラスタリングとはすべての点が集まった状態を頂点として各点がひとつのクラスタになった状態を枝とするような木構造で表現できるクラスタリングのことです。階層型ではないクラスタリングで有名なのは k-means です。 k-means はある点が特定のクラスタに属するという状態が出力なためクラスタ同士に階層がありません。
入力値
一次元の距離尺度ベクトル、若しくは二次元の shape = (クラスタにしたい要素数, クラスタの特徴ベクトル) という形状の配列を指定します。
一次元ベクトルを渡す場合は、クラスタにしたい要素同士 $n$ のおのおのの距離が入った配列を指定する必要があります。そのため長さは必ず $nC_2$ である必要があります。
返り値
返り値は shape = (n-1, 4) の形状の matrix $Z$ です。
$Z$ の第 $i$ 番目の行ベクトルは $i$ 番目のクラスタリングの結果が入っています。
仮に $i$ 番目の行ベクトルを $v$ とします。この時 $v[0]$ と $v[1]$ は新しい $n+i$ 番目のクラスタとして結合される要素の index を表しています。 また $v[2]$ には結合されたクラスタ $v[0], v[1]$ の距離が、 $v[3]$ には結合後のクラスタの要素数がそれぞれ格納されています。
クラスタリングの指定
クラスタリングをどのように実行するか、を制御することができます。これはクラスタリングをする際に何を決めなくてはならないか、を考えるとわかりやすいです。
第一にクラスタリングする際にはクラスタごとの距離を計算する必要があります。これはアルゴリズムが
- 今存在しているクラスタ同士の距離をすべて計算し
- そのなかでもっとも近い物同士を結びつける (linkage)
- 上記を繰り返す
というふうに進んでいく為 step1 でクラスタ間の距離計算を行う必要があるためです。
このクラスタごとの距離の計算を行う関数は「クラスタ化を考えているふたつの集合 $Y, Y'$ の中に存在する2つの要素 $u, v \in Y, Y'$ を入力としてその近さを返す距離尺度 $d: Y \times Y' \to \mathbb{R}$ 」である必要があります。
scipy にはいくつかその関数 (引数としては method で指定します) が用意されていますが、初めは単純な single を使います.
single は以下の数式で表される指標で, クラスタ内の要素の組み合わせすべての中で距離の最小値を返すものです
$$ d(u, v) = \min_{a \in u, b \in v}\left( {\rm dist}(a, b) \right) $$
ここで ${\rm dist}$ は2つのクラスタの要素を与えられた時に実数値を返す関数です. もし入力 y として1次元配列が渡された時は単に絶対値を返す関数となります。
一方で二次元配列 (n, m) が渡された時は m 次元ベクトル二点を入力としてその距離を返す関数 ${\mathbb R}^m \times {\mathbb R}^m \to {\mathbb R}$ を指定する必要があります。(scipy では metrics で指定します)
例
from scipy.cluster.hierarchy import linkage y = [2, 8, 0, 4, 1, 9, 1, 0] # y を特徴ベクトルとしたいので shape = (8, 1) に変換する x = [[d] for d in y] z = linkage(x, method='single', metric='euclid')
この場合だと dist はユークリッド距離になります。各要素の特徴次元が一次元の整数値なのでまあ大体の metric を使っても結果は変わりません。例えばチェビシェフ距離とかでも出力結果は一緒です。
>>> z_abs = linkage(x, method='single', metric='chebyshev') >>> z_abs == z array([[ True, True, True, True], [ True, True, True, True], [ True, True, True, True], [ True, True, True, True], [ True, True, True, True], [ True, True, True, True], [ True, True, True, True]])
つぎにこの出力値を解釈していきます。
>>> z
array([[ 4., 6., 0., 2.],
[ 2., 7., 0., 2.],
[ 0., 8., 1., 3.],
[ 9., 10., 1., 5.],
[ 1., 5., 1., 2.],
[ 3., 11., 2., 6.],
[12., 13., 4., 8.]])
まず初めの行について考えてみます。
>>> z[0] array([4., 6., 0., 2.])
index 0, 1 の値を見ると 4 番目と 6 番目の要素を結合していることがわかります(値はともに 1 です). それらの距離は 0 となっています. 要素がともに同じ値であるので明らかです。そしてこれらの要素は $8 + 0 = 8$ 番目のクラスタに割り当てられました。
>>> z[2] array([0., 8., 1., 3.])
つぎに第2行目をみてみます。ここではクラスタ 0, 8 を結合しています。 クラスタ0 は即ち第0番めの要素でこれは 2 です。一方クラスタ8は8番目の要素(すなわち第1行目で出来たクラスタ)を指しているのでこれらの値はどっちも1でした。
2 と (1, 1,) のユークリッド距離の最小値は 1 なので距離は1です。そしてまとめられたクラスタは要素数3になり, 8 + 2 = 10 番目のクラスタに割り当てられました。
こんな感じにもっとも距離の近いクラスタを集結させていくのが linkage で行われている hierarchical/agglomerative clusteringというものです。
method の種類
method は"single" 以外にもいくつかの種類が用意されています.
https://docs.scipy.org/doc/scipy/reference/generated/scipy.cluster.hierarchy.linkage.html
single や complete といった最小値、最大値を取るものは特徴の中に一つでも近い(遠い)ものがあるときに極端なクラスタを作りがちなのでノイズや変数間のスケールに左右されてしまいます。一般には ward 法が外れ値やスケールに堅牢であるとされているようです。1
Seaborn.clustermap
scipy の linkage の理解が出来たところで seaborn.clustermap の方に戻りましょう. 主な引数は以下のようになっています.
基本
data: pandas.DataFramemethod: default は"average". linkage のクラスタ間の距離尺度を変えたい時はこの値を変更します。scipy.cluster.hierarchy.linkageのmethodと役割は同じです。metric: default は"euclidien". 要素間の距離尺度を変えたいときはこの値を変更します。scipy.cluster.hierarchy.linkageと役割は同じです。 例えば予測値同士の相関に関してクラスタにしてほしいなーという時はmetric="correlation"とかにすると相関ごとにクラスタにしてくれるので良い様な気がします。z_score: 数値が代入されるとその軸方向で 平均 0 分散 1 に正規化します。ずなわち 0 の時が行の方向で 1 の時が列方向です。行方向に正規化するのは画像データとかが該当するでしょうか(各画像ごとに平均ゼロ分散1に揃えたい)。普通の集計データの時は列方向を使うことが多いと思います(列方向に正規化すると各列の値がどれだけ平均からずれているかという無次元情報に変換されるため、特徴ごとのスケールなどを考慮しなくて済むからです)。standard_scale: 数値が代入されるとその軸方向を $[0, 1]$ の範囲に圧縮(拡大)します。分散を1にしないスケーリングです。
ちょっとテクいことをする時
{row,col}_linkage: 行(列)の linkage を指定できます。デフォルトの挙動では行と列に同じ method 及び metric での linkage を行います。違うロジックを使いたいーという時には行の linkage, 列の linkage を予めscipy.cluster.hierarchy.linkageで計算しその結果を渡すとそのクラスタで linkage グラフを描写してくれます。
では実際にいろいろ試してみます。まずは average method でクラスタリングしてみます。
g = sns.clustermap(df_iris, method='average', cmap='viridis') g.ax_col_dendrogram.set_title('Average Euclidean') g.savefig('average_euclid.png', dpi=150)
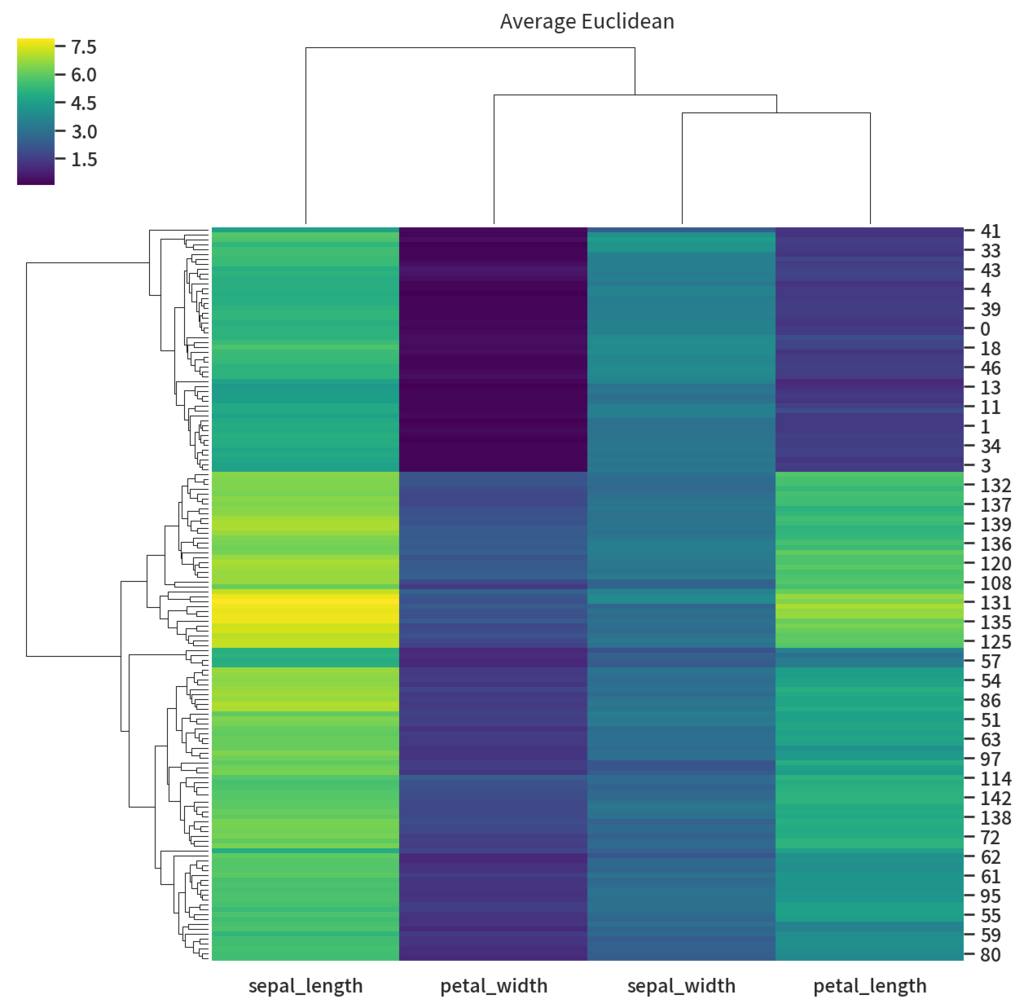
これでもいいんですが少し列間の類似度に差があまり無いように表示されています。これは列に対してユークリッド距離で距離を測っていてかつ正規化などをしていない為に列の値の平均値のズレに引っ張られている為です。
ちょっといたずらで petal_witdh の行に 100足してみましょう
df_fix = df_iris.copy() df_fix['petal_width'] += 100 g = sns.clustermap(df_fix, method='average', cmap='viridis') g.ax_col_dendrogram.set_title('Average Euclidean') g.savefig('fixed_average_euclid.png', dpi=150)
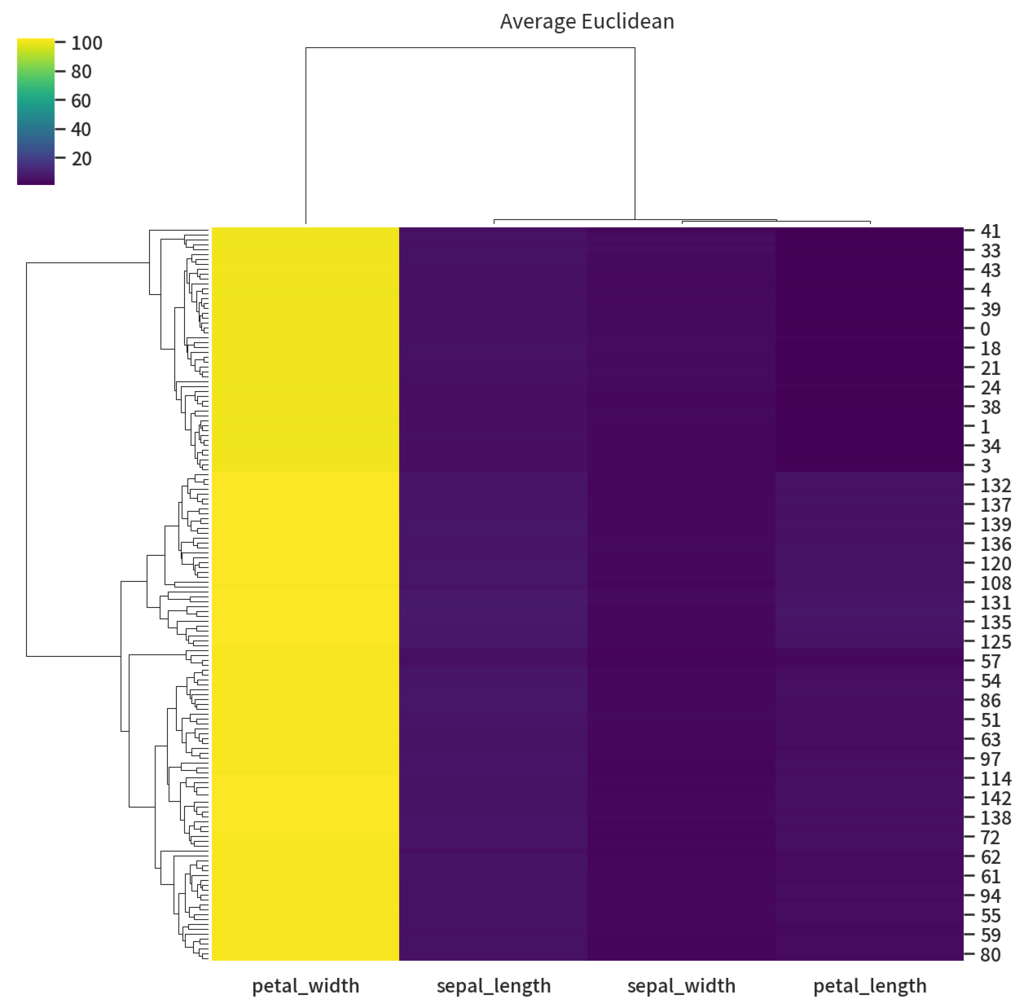
petal_width だけ仲間はずれになってしまいました。これはユークリッド距離を使っているために元データと比べ列比較の際に各要素の差分が 100 増えたことが原因です。
では次は距離をユークリッド距離ではなく相関係数にしましょう。相関係数の式はその系列の平均で引いた後に要素ごとに積を行うので平均を 100 増やしても問題ないはずです。
g = sns.clustermap(df_fix, method='average', metric='correlation', cmap='viridis') g.ax_col_dendrogram.set_title('Average Correlation') g.savefig('fixed_average_correlation.png', dpi=150)

列のクラスタはいい感じになりました! でもちょっと色が意味をなしていないですね。というわけで今度は列方向に正規化します。
g = sns.clustermap(df_fix, method='average', metric='correlation', z_score=1, cmap='viridis') g.ax_col_dendrogram.set_title('Average Correlation') g.savefig('fixed_and_normalized_average_correlation.png', dpi=150)

だいぶ見栄えも良くなりました。ちょっとあれな点があるとすると行方向にも相関を使っている所が微妙かも知れません。 というわけで行にはコサイン類似度を使ってクラスタリングをやるようにしてみます。
# 列方向は相関で col_link = linkage(df_iris.values.T, method='average', metric='correlation') # 行方向はユークリッド距離でクラスタ化 z = (df_fix.values - df_fix.values.mean(axis=0)) / df_fix.values.std() row_link = linkage(z, method='ward', metric='euclidean') g = sns.clustermap(df_fix, col_linkage=col_link, row_linkage=row_link, z_score=1, cmap='viridis') g.ax_col_dendrogram.set_title('Average-Corr Ward-Euclidiean') g.savefig('average-corr__ward-euclidean.png', dpi=150)
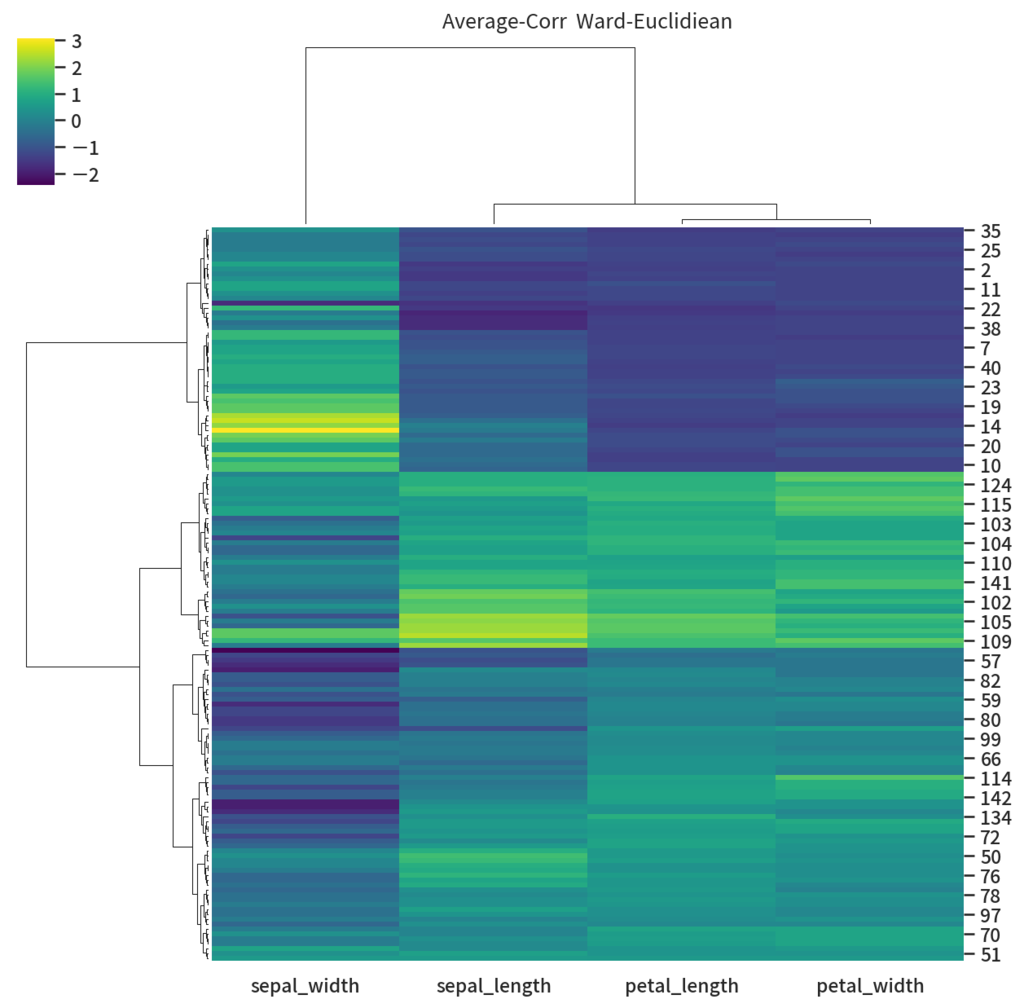
先よりは sepal_width の右上あたりに3あたりを取るクラスタっぽいものができるようになりました。
気をつけたほうが良さそうなこと
以上色々と試してきましたが、実際に可視化をする際に気をつけたほうが良さそうなポイントについて考えてみます。
scaling
今回の iris のような基本的なテーブルデータでは列ごとに意味を持っている場合が多いです。
そのためスケーリングを行うとするならば列方向の指定, 即ち z_score=1 や standard_scale=1 を指定するのが良さそうです。
また初めからスケーリングしてしまうと列ごとの値の大小がわからなくなってしまうため、列ごとの大きさの情報がわかっていない場合はスケーリングせずプロットしてみる、というのもありかも知れません。
アルゴリズムの選定
metric については列に対してはユークリッド距離ではなく相関係数を使ったほうが良いでしょう。ユークリッド距離を使うのであれば、列ごとのスケーリングもしくは平均値を均すような前処理を行うべきです。
method については色々と試してみましたが metric さえちゃんと選んでいれば average や ward など選んでおけば大体事足りそうです。2
一方で "single" や "double" のような最小値/最大値しか見ないロジックを指定すると出力結果が極端になりがちな印象を受けました。のでちょっといつ使ったらいいのかなーという気分です。
またテーブルデータでは列方向には相関 correlation を使いたい場面が多いと思いますが行方向 (レコードごとの距離測定) にも相関でいいのか?という純粋な疑問があります。3 なのでちょっと面倒ですが時間が有る場合は自分で linkage を計算してやるのがよいように思えます。