scikit-learn 準拠の予測モデルのつくりかた
機械学習で色々やっていると、いろいろなモデルを複合したアンサンブルモデルなど、自分で新しい予測モデルを作りたい場合があります。
その場合自分でいちから作り上げても良いのですが、そうやって作ったモデルは、たとえば scikit-learn のパラメータ最適化モジュールである GridSearch や RandomSearch を利用することができなくて、少々不便です。
この際に scikit-learn の定義にしたがってモデルを定義すればうまく連携がとれて効率的です。以下では scikit-learn 準拠の予測モデルをどうやって作ればよいか、その際の注意点や推奨事項を取り上げます。
- 参考
- Creating your own estimator in scikit-learn http://danielhnyk.cz/creating-your-own-estimator-scikit-learn/
Scikit Learnの予測モデル
はじめに、作成するモデルのタイプを選びます。scikit-learn ではモデルは以下の4つのタイプに分類されています。
Classifer- ex). Naive Bayes Classifer などの分類モデル
Clusterring- ex). K-mearns 等のクラスタリングモデル
Regressor- ex). Lasso, Ridge などの回帰モデル
Transformer- ex). PCA などの変数の変換モデル
それぞれのモデルに対して Mixin が定義されていて, BaseEstimator と同時にそれも継承することが推奨されています。
BaseEstimator, 各Mixinのコードは以下を参照してください https://github.com/scikit-learn/scikit-learn/blob/14031f6/sklearn/base.py
予測モデルのコンストラクタでの制約
予測モデルのコンストラクタには以下の制約が存在します。
__init__で呼ばれるすべての引数は初期値を持たなくてはいけません。- 入力変数の確認は
__init__内部では行いません。fitが呼ばれたときに行うようにします。 __init__でのすべての引数は作成されたインスタンスの属性と同じ名前を持つことが推奨されます。(たとえば引数hogeをself.huga = hogeのように与えることはだめです。)__init__ではデータを与えません。fitメソッドで初めて与えるようにします。
Fit method
fit メソッド内ではデータの加工及びパラメータの確認を行います。
この部分以外でデータを取り扱うのは非推奨です(繰り返しになりますが、例えば __init__内部でデータをあたえてnormalizeする等です)。
get_params & set_params
get_params と set_params は BaseEstimator によって定義されている関数です。これをオーバーライドするのは推奨されません。
予測値の取扱
返すベクトルをインプットしなくても良い場合が存在します。(例えばクラスタリングのときなどです)。その場合でも、実装上の問題から、予測値yを引数に定義することが必要です。( y=None で定義することが推奨されます)。こうすることで GridSearch に予測器を与えることが可能になります。
score と gridsearch
グリッドサーチにかけるためには、必要であれば score メソッドをオーバーライドする必要があります。 なぜならばグリッドサーチでは「どのモデルが最も良いのか」を判断する必要があり、その基準となる score メソッドが必要だからです。
score メソッドは 数字が大きいほど良いモデル というルールがあります。 したがって最小化問題が目的関数となっているモデルでは、それにマイナスをつけたものを score として登録する必要があります。 (LassoやRidge回帰のような, 二乗ロス関数を損失関数に持つモデルを gridsearch にかけると、モデルのスコアが負になって表示されるのはこのためです。) 後述する Mixin クラスには score メソッドがすでに定義されているので, Mixin クラスのデフォルトの score メソッドを用いる場合には自分で定義する必要はありません。
サンプル
以上を踏まえた予測器を定義していきます。今回は [訓練データの平均値 + int_val] よりも大きいか小さいかをboolで返す分類器を作成します。分類器なので BaseEstimator と同時に ClassiferMixin も継承しています。
# coding: utf-8 __author__ = "nyk510" from sklearn.base import BaseEstimator, ClassifierMixin from sklearn.model_selection import RandomizedSearchCV import numpy as np class SampleClassifer(BaseEstimator, ClassifierMixin): """ 分類器のサンプル """ def __init__(self, int_val=0, sigma=.5, hogevalue=None): """ 分類器のコンストラクタ 全部の引数に初期値を与えること !! :param int int_val: :param float sigma: :param str hogevalue: """ self.int_val = int_val self.sigma = sigma self.hogevalue = hogevalue self.huga = hogevalue # 引数とインスタンス変数の名前が違っている. 非推奨. def fit(self, X, y=None): """ データへのフィッティングを行うメソッド。 すべての加工, パラメータの確認はここで定義する。 Note: `assert`よりも`try/exception`を用いるほうが本当は良い. けどめんどうなのでassert使ってます :param numpy.ndarray X: 2-D array. 訓練特徴 :param numpy.ndarray y: 1-D array. ターゲット変数 :return: self :rtype: SampleClassifer """ assert(isinstance(self.int_val, int) or isinstance(self.int_val, np.int64)), "int_valは整数値でないと駄目です. " self.treshold_ = (sum(X)/len(X)) + self.int_val return self # return selfするのが慣習 def _meaning(self, x): """ 平均値よりも大きければTrueを返す分類 :rtype: bool """ if x > self.treshold_: return True else: return False def predict(self, X, y=None): """ 予測を行う :param numpy.ndarray X: 特徴量. 2-D array :param numpy.ndarray y: ターゲット変数. 分類問題なので使わないけど`y=None`でおいておく :return: 1-D array :rtype: np.ndarray """ try: getattr(self, "treshold_") except AttributeError: raise RuntimeError("モデルは訓練されていません") return ([self._meaning(x) for x in X]) def score(self, X, y=None): """ モデルの良さを数値化する なんでもいいけれど、大きい方が良くて、小さいほうがだめ。 今回は平均以上の値がいくつあるかをスコアとして定義する。 :return: 平均値の値よりも大きい数 :rtype: int """ return (sum(self.predict(X)))
ところでこの ClassiferMixin にある Mixin とは何でしょうか。wikipediaを引用すると
mixin とはオブジェクト指向プログラミング言語において、サブクラスによって継承されることにより機能を提供し、単体で動作することを意図しないクラスである。 Mixin - Wikipedia
Mixin は継承することで初めて機能を提供できるクラスを指しています。Scikit-Learnの ClassiferMixin のコードを見ると以下が定義されています。
class ClassifierMixin(object): """Mixin class for all classifiers in scikit-learn.""" _estimator_type = "classifier" def score(self, X, y, sample_weight=None): """Returns the mean accuracy on the given test data and labels. In multi-label classification, this is the subset accuracy which is a harsh metric since you require for each sample that each label set be correctly predicted. Parameters ---------- X : array-like, shape = (n_samples, n_features) Test samples. y : array-like, shape = (n_samples) or (n_samples, n_outputs) True labels for X. sample_weight : array-like, shape = [n_samples], optional Sample weights. Returns ------- score : float Mean accuracy of self.predict(X) wrt. y. """ from .metrics import accuracy_score return accuracy_score(y, self.predict(X), sample_weight=sample_weight)
自分の名前(_estimator_type)とscore関数のデフォルトが定義されています。
score関数内ではインスタンスメソッドのself.predict(X)が呼ばれているので、このクラスはBaseEstimatorを継承してpredictを持っているインスタンスに継承されて初めて意味があるものだとわかります。
python では __init__ メソッドを定義せずにインスタンスメソッドのみを定義することで、新しく Mixin クラスを作ることが可能です。
つぎに、適当にデータを作成してこのモデルに対してランダムサーチをしてみましょう。
x_train = np.random.normal(.1, 4, size=100) x_test = np.random.normal(-.1, 4, size=20) model_params = { "int_val": [-10, -1, 0, 1, 2], "sigma": np.linspace(-1, 1, 100) } clf = SampleClassifer() random_search = RandomizedSearchCV(estimator=clf, param_distributions=model_params) random_search.fit(x_train)
score関数はTrueの数が多いほど大きいという風に定義していますから、int_valが一番小さい値(すなわち-10)の予測器がbest_estimator_となっているはずです。
(本当はRandomSearchなので -10 が選択されているとは限らない, ということに今気が付きました……ほんとうはGridSearchすべきでしたね)1
random_search.best_params_
{'int_val': -10, 'sigma': 0.51515151515151536}
ちゃんと -10 が選ばれていることが分かります。
-
int_valのとりうる値の数は5なので, 1 - (4/5)**10 ~ 0.892 より, -10 が1回でも選ばれる確率はだいたい 89.2%です↩
matplotlibで高校生っぽいグラフを描く
高校の時によくみたグラフをmatplotlibで書いてみたくなったので、やってみました。
練習として初めてデコレーターを使って、プロットしたオブジェクトをいじって軸をあれこれするようにしてみました。
機能としてあるのは知っていたのですが、使ってみるとデコレーターって意外と便利ですね。やっぱり食わず嫌いは良くないなと思いました。
作れる画像はこんな感じです。


意外と苦労するのが軸の矢印を作るところで、画像の縦横比が変わった時にもちゃんと特定の比率の長方形に頂点を持つ矢印を持つようにしてあります。さっくり適当なグラフを書くのには使えるかも。
DCGANをChainerで実装
DCGANとは
DCGANはランダムなベクトルから画像を生成するGeneratorと、画像が与えられた時にその画像がGeneratorによって作られたものなのか、本当の画像なのかを判定する関数Discriminatorでなりたっています。
DCGANのアイディア
この2つの関数を相互に競わせるように更新することで、画像を生成するGeneratorをもっともらしいものへと更新します。
Generatorは、できるだけDiscriminatorに偽物だと判定されないように、重みを更新します。
反対に、Discriminatorは、できるだけGeneratorの画像を本物だと判定しないように、重みを更新します。
しくみとしてはこれだけで構成されていて、DCGANではGeneratorに逆Convolution(Deconvolution)を、DiscriminatorにConvolutionを用います。またConvolutionを行う際、BatchNormalizationを行うことで、高速な学習を可能にします。
実装する
今回はChainerを用いて、DCGANを実装しました。コードはgithubに置いています。https://github.com/Nyker510/hobby
僕のローカル環境ではGPUを使うことができないので、cpuを用いたコードになっていますので、そのまま使うととても遅いのでそれだけは注意してください。
構成としてはGenerator,Discriminatorを定義しているdcgan.pyとデータを与えて最適化していくtrainer.pyで構成されています。
dcgan.py
まずはGeneratorから
class Generator(Chain): '''ランダムなベクトルから画像を生成する画像作成機 ''' def __init__(self, z_dim): super(Generator, self).__init__( l1=L.Linear(z_dim, 3 * 3 * 512), dc1=L.Deconvolution2D(512, 256, 2, stride=2, pad=1,), dc2=L.Deconvolution2D(256, 128, 2, stride=2, pad=1,), dc3=L.Deconvolution2D(128, 64, 2, stride=2, pad=1,), dc4=L.Deconvolution2D(64, 1, 3, stride=3, pad=1), # Convolution, Deconvolutionともに値は画像の大きさに合わせて変化させる # 必要がある。 # 今回はMNISTをターゲットにするので、元の大きさは28×28 # 元の512チャンネルから1チャンネル(MNISTは白黒なためチャンネルが無い)に変換するまでに # 3→4→5→10→28となるようにstride,pad,windowの大きさを選んでいる # bn0 = L.BatchNormalization(6*6*512), bn1=L.BatchNormalization(512), bn2=L.BatchNormalization(256), bn3=L.BatchNormalization(128), bn4=L.BatchNormalization(64), ) self.z_dim = z_dim def __call__(self, z, test=False): h = self.l1(z) # 512チャンネルをもつ、3*3のベクトルに変換する h = F.reshape(h, (z.data.shape[0], 512, 3, 3)) h = F.relu(self.bn1(h, test=test)) h = F.relu(self.bn2(self.dc1(h), test=test)) h = F.relu(self.bn3(self.dc2(h), test=test)) h = F.relu(self.bn4(self.dc3(h), test=test)) x = self.dc4(h) return x
Generatorはランダムなz_dim次元ベクトルを受け取って画像として返す関数です。今回実験対称としてMNISTの白黒画像を用いることを想定しているので、最後画像として返すDeconvolution関数を(28,28)次元のチャンネル数1と鳴るように、stride,pad,windowの大きさを選んでいます。
なので、違う大きさの画像でやりたいという場合や、RGBのある画像(チャンネル数が3)でやりたいというときは、それに合わせて変化させる必要があります。
(例えばRGBでやりたいのであれば、 dc4=L.Deconvolution2D(64, 3, 3, stride=3, pad=1)です)
これと反対方向に、即ち画像を受け取って2値に写像する関数として、Discriminatorも定義します。
trainer.py
trainer.pyでは実際に最適化を行っていきます。
大切なところは、lossをどのように与えるか、の部分です。
z = np.random.uniform(-1, 1, (batchsize, self.z_dim)) z = z.astype(dtype=np.float32) z = Variable(z) x = self.gen(z) y1 = self.dis(x) # 答え合わせ # ジェネレーターとしては0と判別させたい(騙すことが目的) loss_gen = F.softmax_cross_entropy( y1, Variable(np.zeros(batchsize, dtype=np.int32))) # 判別機としては1(偽物)と判別したい loss_dis = F.softmax_cross_entropy( y1, Variable(np.ones(batchsize, dtype=np.int32))) # load true data form dataset idx = perm[i * batchsize:(i + 1) * batchsize] x_data = self.X[idx] x_data = Variable(x_data) y2 = self.dis(x_data) # 今度は正しい画像なので、0(正しい画像)と判別したい loss_dis += F.softmax_cross_entropy( y2, Variable(np.zeros(batchsize, dtype=np.int32)))
仮定として、Discriminatorの二次元の出力の内、0次元目を正しい画像である確率、1次元目を間違った画像(Generatorによって作成された偽画像)である確率であるとします。
まずzを適当なランダムベクトルとして設定した後、それをGeneratorに渡すことで画像xを作成します。その後、xをDiscriminatorに渡して二次元のベクトルy1を得ます。
Generatorとしては、Discriminatorを騙すことが目的です。
言い換えると、この画像xをDiscriminatorに渡した時に「本物だ!」と思わせたいのです。したがって全てのy1が0次元目の値が大きくなって欲しいので、ロスとしてy1と0とのSOFTMAXを与えます。
反対にDiscriminatorとしては、この画像xをちゃんと偽物であると判定しなくてはなりません。
すなわち1次元目が大きい値を取るようになって欲しいので、Discriminatorのロストしてはy1と1とのSOFTMAXを与えます。
そしてつぎに,Discriminatorに正しい画像も与えてあげて、これをちゃんと正しい画像(つまり0)と判定してほしいので、y2と0とのSOFTMAXをロスに加えます。
実験結果
MNISTの画像を使って実験を行います。本当は0~9でやれば一番良いのですが、GPUが使えない環境なので無限に時間がかかって終わりません。ですので泣く泣く、0の画像だけを用いて実験を行いました。
training中、epochが終わるごとにランダムなベクトルから画像をGeneratorによって作成してプロットさせていて、その画像を示します。同時にDiscriminatorが出力した「この画像が本物である確率」も画像の上に表示しています。
1 epoch目

ただのノイズです。
100 epoch目

だいぶ画像っぽくなってきました。がしかしDiscriminatorには正しい画像だとは思われていないようです。
500 epoch目

かなりしっかりと0を作るようになっています。正しい画像である確率が0.3程度になるなど、Discriminatorも判定に苦労している様子が伺えます。
1000 epoch目

500epoch目に比べて白黒の部分がはっきりとしてきました。
まとめ
今回はMNISTの0の画像だけというシンプルなタスクですが、学習が進むに連れて明確な輪郭をもって画像を出力する様子が確認できました。GPU環境であれば、更に高次の特徴をもった画像(例えばキャラクターのイラストなど)でも作成できるようなので、画像の自動生成技術として様々な用途で使えるかもしれません。
参考文献
PRML 10章の変分ベイズ法の実装
線形回帰に対する変分法を用いた計算について実装してみました。
コードは以下から
github.com
変分ベイズではまず、隠れ変数も含めた完全データに対して同時分布を定義します。その後隠れ変数の分布が複数の関数の積として近似して表すことができる、という仮定を導入します。
このことによって、完全データに対する同時分布を、目的の隠れ変数以外の事後分布で周辺化したものが、新しい近似事後分布となり、それをすべての隠れ変数に対して繰り返して計算していくことにより、より尤度の高い隠れ変数の事後分布を計算していく、という手法です。
今回は次のグラフィカルモデルで表すことができる確率モデルに対して、変分ベイズ方を適用します。
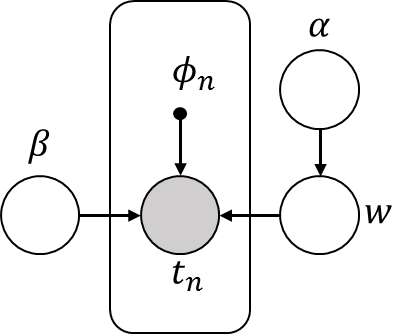
PRML10.3と似ていますが、実際のラベルデータが生成される際の精度パラメータβに対しても分布を定義している部分が異なっています。βに分布をもたせるとどのぐらい変化があるのかということを確かめたかったので、βにも分布をもたせました。
データとしては正しいsin関数にsigma=.5のガウスノイズを付加したデータを用います。
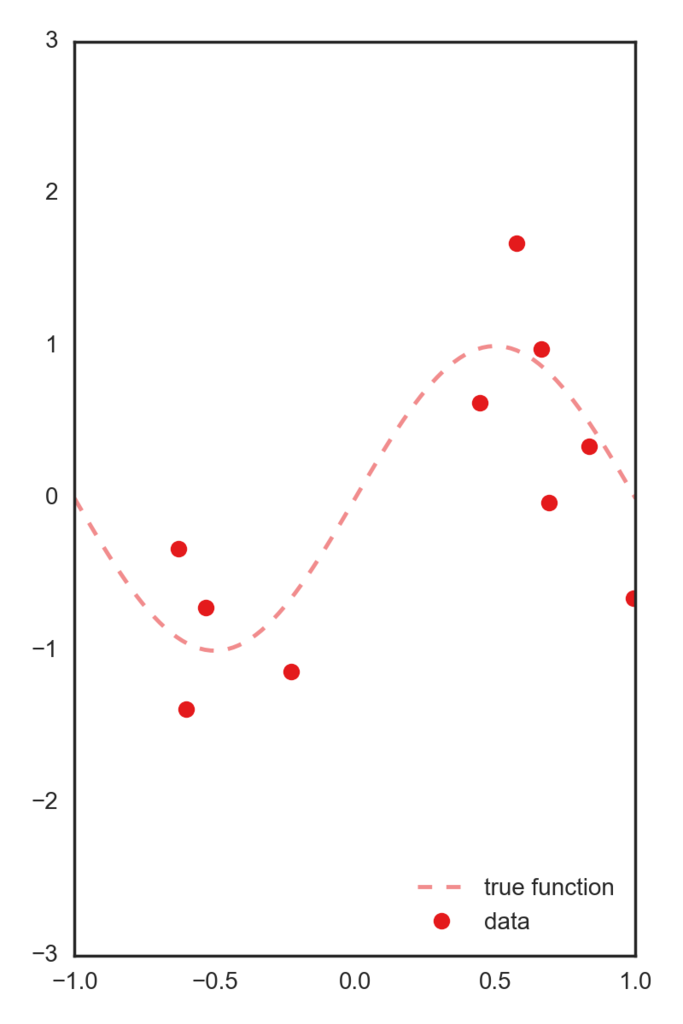
この一次元特徴を、[-1~1]を5個に分割した中心を持つガウス基底によって5次元空間に写像したデータを特徴量として用います。 以下コードです。
import numpy as np import matplotlib.pyplot as plt import pandas as pd np.random.seed(71) def t_func(x): """正解ラベルを作る関数 x: numpy array. return t: target array. numpy.array like. """ t = np.sin(x * np.pi) # t = np.where(x > 0, 1, -1) return t def plot_target_function(x, ax=None, color="default"): """target関数(ノイズなし)をプロットします """ if ax is None: ax = plt.subplot(111) if color is "default": color = "C0" ax.plot(x, t_func(x), "--", label="true function", color=color, alpha=.5) return ax def phi_poly(x): dims = 3 return [x ** i for i in range(0, dims + 1)] def phi_gauss(x): bases = np.linspace(-1, 1, 5) return [np.exp(- (x - b) ** 2. * 10.) for b in bases] def qw(alpha, phi, t, beta): """ wの事後分布を計算します。 変分事後分布はガウス分布なので決定すべきパラメータは平均と分散です w ~ N(w| m, S) return ガウス分布のパラメータ m, S """ S = beta * phi.T.dot(phi) + alpha * np.eye(phi.shape[1]) S = np.linalg.inv(S) m = beta * S.dot(phi.T).dot(t) return m, S def qbeta(mn, Sn, t, Phi, N, c0, d0): """ betaの変分事後分布を決めるcn,dnを計算します 変分事後分布はガンマ分布なので決定すべきパラメータは2つです beta ~ Gamma(beta | a, b) return ガンマ分布のパラメータ a,b """ cn = c0 + .5 * N dn = d0 + .5 * (np.linalg.norm(t - Phi.dot(mn)) ** 2. + np.trace(Phi.T.dot(Phi).dot(Sn))) return cn, dn def qalpha(w2, a0, b0, m): """ alphaの変分事後分布を計算します。 変分事後分布はガンマ分布ですから決定すべきパラメータは2つです alpha ~ Gamma(alpha | a, b) return a, b """ a = a0 + m / 2. b = b0 + 1 / 2. * w2 return a, b def fit(phi_func, x, update_beta=False): xx = np.linspace(-2, 2., 100) if phi_func == "gauss": phi_func = phi_gauss elif phi_func == "poly": phi_func == phi_poly else: if type(phi_func) == "function": pass else: raise Exception("invalid phi_func") Phi = np.array([phi_func(xi) for xi in x]) Phi_xx = np.array([phi_func(xi) for xi in xx]) # 変分事後分布の初期値 N, m = Phi.shape mn = np.zeros(shape=(Phi.shape[1],)) Sn = np.eye(len(mn)) beta = 10. alpha = .1 a0, b0 = 1, 1 c0, d0 = 1, 1 pred_color = "C1" freq = 2 n_iter = 3 * freq n_fig = int(n_iter / freq) fig = plt.figure(figsize=(3 * n_fig, 4)) data_iter = [] data_iter.append([alpha, beta]) for i in range(n_iter): print("alpha:{alpha:.3g} beta:{beta:.3g}".format(**locals())) mn, Sn = qw(alpha, Phi, t, beta) w2 = np.linalg.norm(mn) ** 2. + np.trace(Sn) a, b = qalpha(w2, a0, b0, m) c, d = qbeta(mn, Sn, t, Phi, N, c0, d0) alpha = a / b if update_beta: # betaが更新される beta = c / d data_iter.append([alpha, beta]) if i % freq == 0: k = int(i / freq) + 1 ax_i = fig.add_subplot(1, n_fig, k) plot_target_function(xx, ax=ax_i) ax_i.plot(x, t, "o", label="data", alpha=.8) m_line = Phi_xx.dot(mn) sigma = (1. / beta + np.diag(Phi_xx.dot(Sn).dot(Phi_xx.T))) ** .5 ax_i.plot(xx, m_line, "-", label="predict-line", color=pred_color) ax_i.fill_between(xx, m_line + sigma, m_line - sigma, label="Predict 1 sigma", alpha=.2, color=pred_color) ax_i.set_title( "n_iter:{i} alpha:{alpha:.3g} beta:{beta:.3g}".format(**locals())) ax_i.set_ylim(-2, 2) ax_i.set_xlim(-1.5, 1.5) if i == 0: ax_i.legend(loc=4) fig.tight_layout() return fig, data_iter
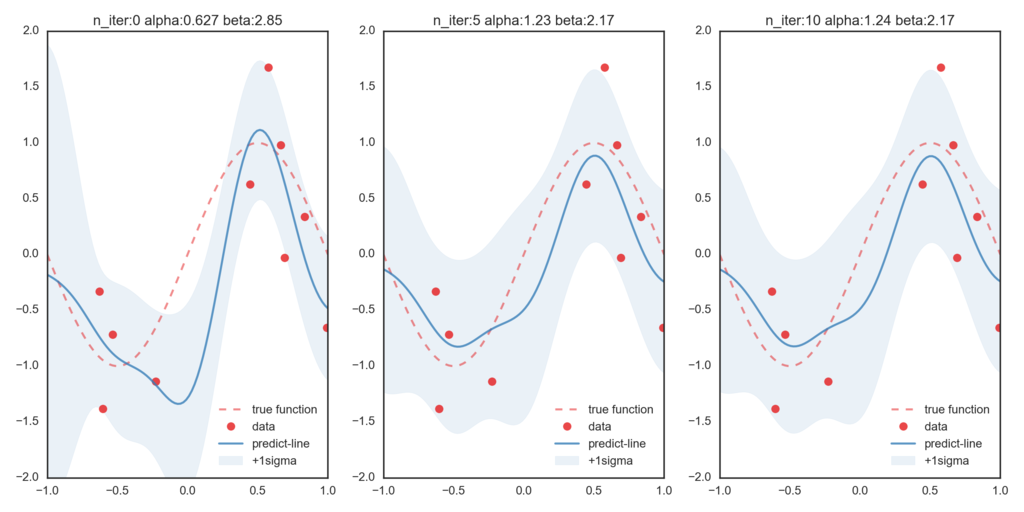
まずはβを更新せずハイパーパラメータとして与えて、変分ベイズを適用します。(β=10で固定)
一番左が初期値での最尤推定値となっていて、右に行くほど変分ベイズ法によって更新された分布情報に基づいた予測値になっています。今回与えたβが、実際の精度パラメータよりも大きな値を取っているために、データ点に過剰にフィットしていることが伺えます。
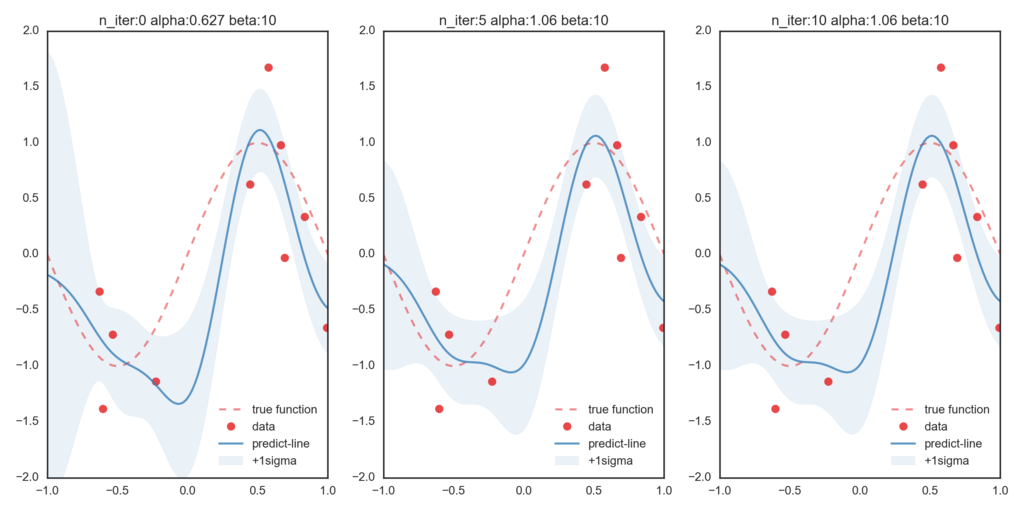
つぎにβを変分ベイズ法で更新したのが次のグラフです。比べてみると、βが更新されることによって正しい関数に近づいていることが確認できます。