PRML 10章の変分ベイズ法の実装
線形回帰に対する変分法を用いた計算について実装してみました。
コードは以下から
github.com
変分ベイズではまず、隠れ変数も含めた完全データに対して同時分布を定義します。その後隠れ変数の分布が複数の関数の積として近似して表すことができる、という仮定を導入します。
このことによって、完全データに対する同時分布を、目的の隠れ変数以外の事後分布で周辺化したものが、新しい近似事後分布となり、それをすべての隠れ変数に対して繰り返して計算していくことにより、より尤度の高い隠れ変数の事後分布を計算していく、という手法です。
今回は次のグラフィカルモデルで表すことができる確率モデルに対して、変分ベイズ方を適用します。
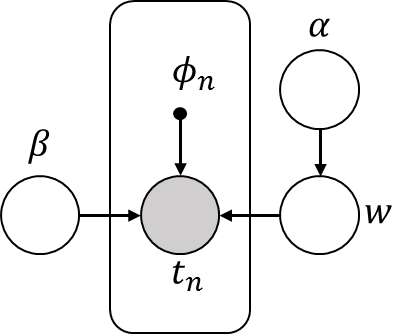
PRML10.3と似ていますが、実際のラベルデータが生成される際の精度パラメータβに対しても分布を定義している部分が異なっています。βに分布をもたせるとどのぐらい変化があるのかということを確かめたかったので、βにも分布をもたせました。
データとしては正しいsin関数にsigma=.5のガウスノイズを付加したデータを用います。
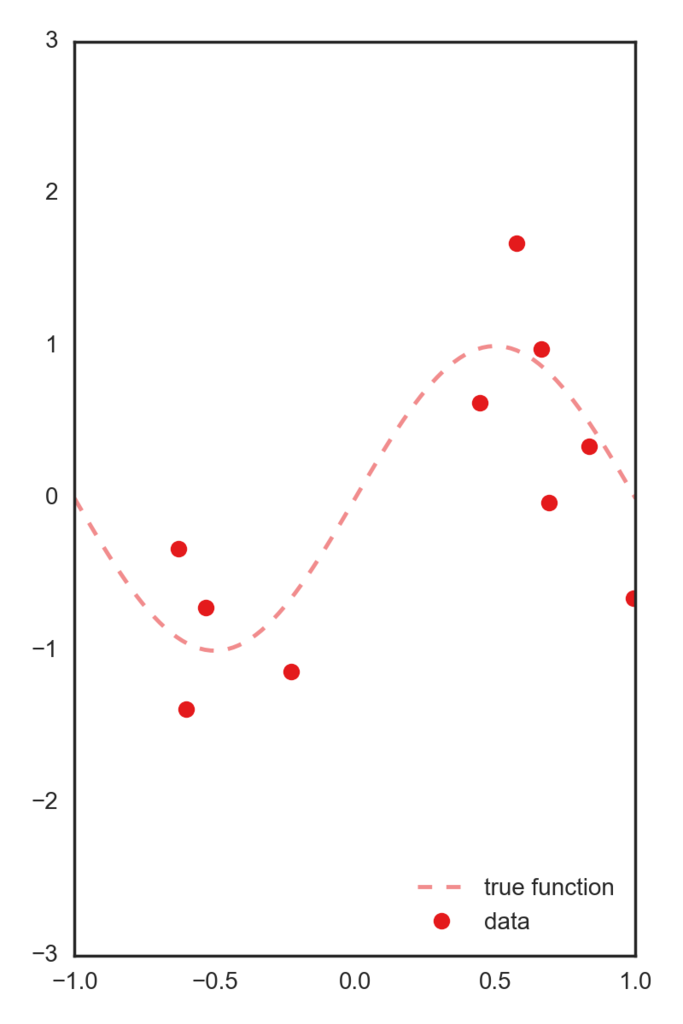
この一次元特徴を、[-1~1]を5個に分割した中心を持つガウス基底によって5次元空間に写像したデータを特徴量として用います。 以下コードです。
import numpy as np import matplotlib.pyplot as plt import pandas as pd np.random.seed(71) def t_func(x): """正解ラベルを作る関数 x: numpy array. return t: target array. numpy.array like. """ t = np.sin(x * np.pi) # t = np.where(x > 0, 1, -1) return t def plot_target_function(x, ax=None, color="default"): """target関数(ノイズなし)をプロットします """ if ax is None: ax = plt.subplot(111) if color is "default": color = "C0" ax.plot(x, t_func(x), "--", label="true function", color=color, alpha=.5) return ax def phi_poly(x): dims = 3 return [x ** i for i in range(0, dims + 1)] def phi_gauss(x): bases = np.linspace(-1, 1, 5) return [np.exp(- (x - b) ** 2. * 10.) for b in bases] def qw(alpha, phi, t, beta): """ wの事後分布を計算します。 変分事後分布はガウス分布なので決定すべきパラメータは平均と分散です w ~ N(w| m, S) return ガウス分布のパラメータ m, S """ S = beta * phi.T.dot(phi) + alpha * np.eye(phi.shape[1]) S = np.linalg.inv(S) m = beta * S.dot(phi.T).dot(t) return m, S def qbeta(mn, Sn, t, Phi, N, c0, d0): """ betaの変分事後分布を決めるcn,dnを計算します 変分事後分布はガンマ分布なので決定すべきパラメータは2つです beta ~ Gamma(beta | a, b) return ガンマ分布のパラメータ a,b """ cn = c0 + .5 * N dn = d0 + .5 * (np.linalg.norm(t - Phi.dot(mn)) ** 2. + np.trace(Phi.T.dot(Phi).dot(Sn))) return cn, dn def qalpha(w2, a0, b0, m): """ alphaの変分事後分布を計算します。 変分事後分布はガンマ分布ですから決定すべきパラメータは2つです alpha ~ Gamma(alpha | a, b) return a, b """ a = a0 + m / 2. b = b0 + 1 / 2. * w2 return a, b def fit(phi_func, x, update_beta=False): xx = np.linspace(-2, 2., 100) if phi_func == "gauss": phi_func = phi_gauss elif phi_func == "poly": phi_func == phi_poly else: if type(phi_func) == "function": pass else: raise Exception("invalid phi_func") Phi = np.array([phi_func(xi) for xi in x]) Phi_xx = np.array([phi_func(xi) for xi in xx]) # 変分事後分布の初期値 N, m = Phi.shape mn = np.zeros(shape=(Phi.shape[1],)) Sn = np.eye(len(mn)) beta = 10. alpha = .1 a0, b0 = 1, 1 c0, d0 = 1, 1 pred_color = "C1" freq = 2 n_iter = 3 * freq n_fig = int(n_iter / freq) fig = plt.figure(figsize=(3 * n_fig, 4)) data_iter = [] data_iter.append([alpha, beta]) for i in range(n_iter): print("alpha:{alpha:.3g} beta:{beta:.3g}".format(**locals())) mn, Sn = qw(alpha, Phi, t, beta) w2 = np.linalg.norm(mn) ** 2. + np.trace(Sn) a, b = qalpha(w2, a0, b0, m) c, d = qbeta(mn, Sn, t, Phi, N, c0, d0) alpha = a / b if update_beta: # betaが更新される beta = c / d data_iter.append([alpha, beta]) if i % freq == 0: k = int(i / freq) + 1 ax_i = fig.add_subplot(1, n_fig, k) plot_target_function(xx, ax=ax_i) ax_i.plot(x, t, "o", label="data", alpha=.8) m_line = Phi_xx.dot(mn) sigma = (1. / beta + np.diag(Phi_xx.dot(Sn).dot(Phi_xx.T))) ** .5 ax_i.plot(xx, m_line, "-", label="predict-line", color=pred_color) ax_i.fill_between(xx, m_line + sigma, m_line - sigma, label="Predict 1 sigma", alpha=.2, color=pred_color) ax_i.set_title( "n_iter:{i} alpha:{alpha:.3g} beta:{beta:.3g}".format(**locals())) ax_i.set_ylim(-2, 2) ax_i.set_xlim(-1.5, 1.5) if i == 0: ax_i.legend(loc=4) fig.tight_layout() return fig, data_iter
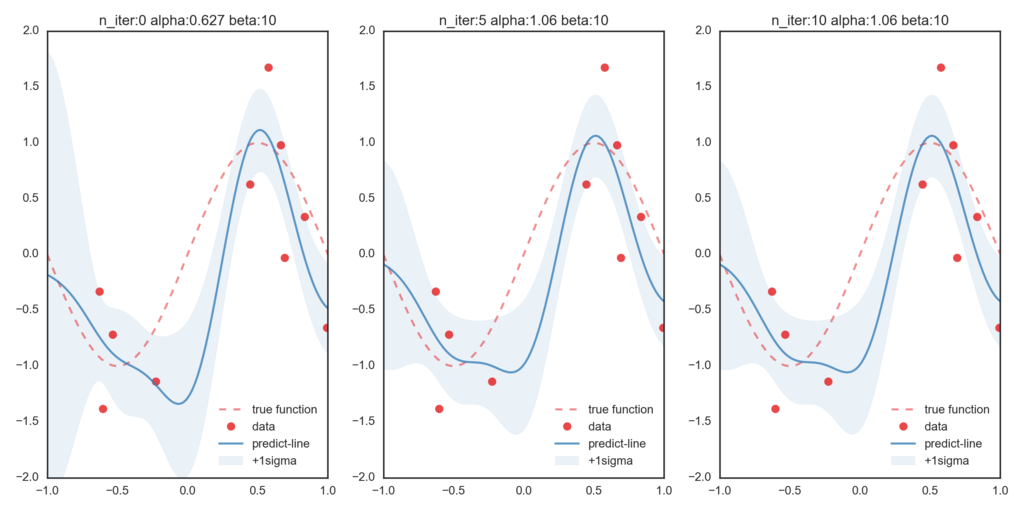
まずはβを更新せずハイパーパラメータとして与えて、変分ベイズを適用します。(β=10で固定)
一番左が初期値での最尤推定値となっていて、右に行くほど変分ベイズ法によって更新された分布情報に基づいた予測値になっています。今回与えたβが、実際の精度パラメータよりも大きな値を取っているために、データ点に過剰にフィットしていることが伺えます。
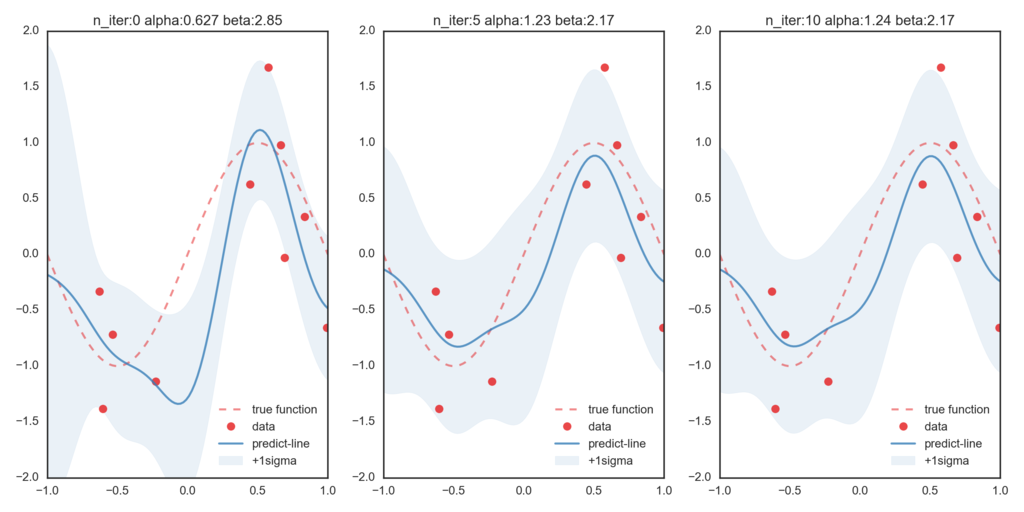
つぎにβを変分ベイズ法で更新したのが次のグラフです。比べてみると、βが更新されることによって正しい関数に近づいていることが確認できます。
format記法の使い道
pythonでは変数の情報を文字列にする方法がいくつかありますが、その中でも僕はformat記法をよく使っています。
特に最近「dictionaryを文字列にしたいなぁ」という場合に、とてもきれいに書くことができるということを発見(というかドキュメントを読んで判明した)ので「こういう場合に便利ですよ」という意味合いでメモしておきます。
Case: dictionaryからstringに変換したい
例えば、機械学習なんかをやろうと思うと、パラメータをディクショナリとして渡す場面がよくあると思います。仮にそのパラメータをparamsとして以下のように定義します。
params = {
'estimator':'svm',
'gamma':0.1,
'eta':100,
}
このパラメータを使って学習した結果を保存しよう、となると「このパラメータ使いましたよ」という名前で保存したくなります。そういうときにdictionary→stringに変換したくなります。
単純に要素をすべて列挙してそれをつなげる、という風にすると以下のようなプログラムが考えられます。
pstr = '' for key,val in params.items(): if pstr != '': pstr += '_' pstr += key + '-' +str(val) print(pstr) # <<< 'estimator-svm_eta-100_gamma-0.1'
これでいいように感じるのですが、一つ問題があります。
dictionaryは入っている要素の順番を保存していないので、実験を繰り返しているとタイトルの順番が変化してしまう場合があるのです。(実際にあってとても困りました)
'estimator-svm_eta-100_gamma-0.1' これが 'eta-100_estimator-svm_gamma-0.1' こんな風になる場合があるってこと
また先のスクリプトでは辞書の値をすべてstrを用いて文字に変換しています。これも問題があって、例えばfloatをstrを用いて文字にすると桁数の指定ができないのでstr(val)[:2]のようにして長さを揃えたりしなくてはならず、しかもこれをすると四捨五入等も行ってくれないので、まあ言ったらちょっと不便です。
そこで最近はformat記法を用いて文字列に変換するようにしています。
estimator = 'svm' gamma = 0.166 eta = 100 pstr = 'estimator-{0}_gamma-{1:.2f}_eta-{2}'.format(estimator,gamma,eta) print(pstr) # estimator-svm_gamma-0.17_eta-100
{}の中の番号がformatで与えた引数の順番に対応しています。また同時にそれをどういう形でstrにするかも指定できるので、先の例だとgammaを下二桁の少数で、という指定をしているので0.17として出力されています。
これでもだいぶ便利なのですが、コードを書いていて「新しい変数hogeも名前の先頭に追加したいなー」となったとき、先頭にhogeを追加すると、順番がひとつづつずれてしまうので、0,1,2を1,2,3にしなくてはならなくなり、面倒です。
これを回避するためには、最後にhogeを追加して、3番めの引数を文字列の先頭に持ってくる、というformat内部の並びと、文字の対応が取れないコードになってしまいます。ちょっとアホっぽいですね。
hoge = 0.2 estimator = 'svm' gamma = 0.166 eta = 100 pstr = 'hoge-{3:.3f}_estimator-{0}_gamma-{1:.2f}_eta-{2}'.format(estimator,gamma,eta,hoge) print(pstr) # hoge-0.200_estimator-svm_gamma-0.17_eta-100
辞書の展開わたし
これを回避するのが、辞書を展開して渡してあげる、という方法です。(format(**some_dict))
展開した辞書の値をほしいときには{key}としてあげます。具体的には一番最初の例だとこんな感じ。
pstr = 'estimator-{estimator}'.format(**params) print(pstr) # estimator-svm
この書き方の良い点は、stringの中にkeyを書くことになるので、ぱっと見たときにこの文字列が何になるのか、がわかりやすいという点です。
短所として長くなると手で書いているのが面倒になるという点がありますが、それでも読みやすいわかりやすいというメリットは大きいと思います。
しかしこの方法は文字にしたいものが最初からdictionaryになっている場合にしか使えません。
locals()
なのですが、これと組み込み関数のlocals()を用いるとその場合でも楽に変換できます。
localsはその場に定義されている変数を辞書にして返す関数です。なのでこれとformatを組み合わせると、その場にある変数の文字列を先の記法で作ることができます。
val = 100 name = 'some_method' tstr = 'val={val}_name={name}'.format(**locals()) print(tstr) # val=100_name=some_method
0,1,2と指定する場合に比べて、明らかに見やすいですね。
まとめ
localsとか展開渡しとか全然知らずにアホっぽいコード量産していました。ドキュメントはちゃんと読みましょう。
pythonの自作モジュールimportで困ったこと
自作モジュールをimportしたい時
同じ階層にある時はsample.pyであれば
import sample
でやればOKです.
でもあまりファイル数が多くなってくると,一定のまとまりでフォルダに入れて管理したくなります.そういう時は適当なフォルダにスクリプトを入れて,そのフォルダ内に__init__.pyを入れておけば,pythonは再帰的にフォルダ内部の.pyファイルを読み込んでくれるので,importができるようになります.
今回詰まったこと
フォルダ構造は以下のような感じ

sample.py
import numpy as np def sample(x): x = np.random.normal(size=100) print(x.mean())
begin.py
from sample import sample sample()
これをtest_moduleの親ディレクトリにある以下のファイルからimportしようとするとエラーが起こりました.
from test_module import begin begin.sample()
File "/Users/NYer510/python-tutorial/test_module/begin.py", line 2, in <module>
from sample import sample
ImportError: No module named 'sample'
解決法
結論から言うと,encodingを指定すると直りました.(# -- coding: utf-8 --とかのやつ)
理由はわかりません.謎です.
python の Xgboost を Windows で使いたい!
pythonをwindowsでやるなよ、という意見はごもっともですがでもやりたい時だってあるじゃん?なのでやりましょう。
環境
- windows10
- 64bit
必要なものたち
git cloneする
git bash を起動して、xgboostをgithubから手に入れます。
ここで注意するポイントが2つ
git cloneしたあとに submodule を綺麗にして入れ直すこと。- 現在の最新版(2016/06/28現在)のmasterには問題があるようなのでちょっと前の commit に戻すこと。
コマンドとしてはgit checkout 9a48a40.
Error installing xgboost · Issue #1267 · dmlc/xgboost · GitHub
$ git clone --recursive https://github.com/dmlc/xgboost $ cd xgboost $ git checkout 9a48a40 $ git submodule init $ git submodule update
MinGW-W64のインストール
次にMinGW-W64をインストールします。この時に architecture を x84_64 にセットしてインストールします。加えてデフォルトではC:\Program File直下にインストールすることになっていますが、空文字を避けるためにC:\mingw64とか適当にCドライブ直下に新しくフォルダを作り、そこを指定します。
Pathの設定
環境変数設定からPathを追加します。追加するのはさっきインストールしたMingw64の中にあるmingw32-make.exeが入っているフォルダです。C:\mingw64\mingw64\bin とかになるはず。

どうでもいいけどwindows10になって環境変数の編集画面がすごく見やすくなりましたね。個人的には好きです。
コンパイル
make の設定を行ってからコンパイルします。以下のコマンドを先のgit bash 上の、xgboostをコピーした階層で行います
$ cp make/mingw64.mk config.mk $ make -j4
なんかもにゃもにゃ出るので待ちます。
インストール
成功していれば xgboost の階層に xgboost.exe ができているはずです。python パッケージをインストールする場合には python-pakage フォルダで setup.py を実行します。
$ cd python-package $ python setup.py install
結構長いですね。はやくconda install xgboostでwindowsでも入れられるような幸せな世界になって欲しいです。